Mit künstlicher Intelligenz Leben retten
Der Doktorand Fabian Laumer und seine Kolleginnen und Kollegen nutzen maschinelles Lernen, um neue personalisierte 3D-Modelle eines schlagenden menschlichen Herzens zu entwickeln. Ihr Ziel ist es, Fachkräfte im Bereich der Kardiologie zu unterstützen und die Diagnose sowie Behandlung von kardiovaskulären Erkrankungen zu verbessern, welche weltweit die am häufigsten auftretende Todesursache sind.
-
-
(Illustration: Fabian Laumer / D-INFK) -
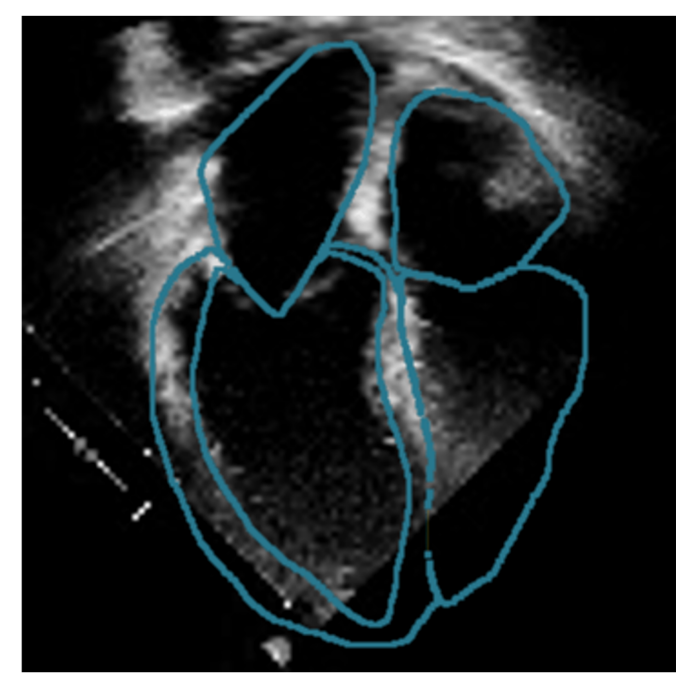
Kommen Personen mit Anzeichen eines Herzinfarkts im Spital an, messen die Ärztinnen und Ärzte verschiedene Parameter, um die Schwere der Symptome einschätzen und eine Behandlungsstrategie festlegen zu können. Einer dieser Parameter ist die «linksventrikuläre Ejektionsfraktion», eine Messung der Herzeffizienz, die bei der Vorhersage einer Pathologie oder der Chancen auf einen Rückfall hilfreich ist: «Nach einem ersten Herzereignis», erklärt Fabian Laumer, «korreliert ein weniger effizientes Herz mit einem früheren Tod. Die Messung dieser Parameter ist jedoch aufwendig und nicht immer präzise.»
Fabian ist Doktorand in der Information and Science Engineering Group von Professor Buhmann. Dort konzipiert er Machine-Learning-Tools zur Modellierung menschlicher Herzen. Im Rahmen seiner Masterarbeit entwickelte er einen Algorithmus, der Kardiolog:innen dabei unterstützt, einen Herzinfarkt von anderen Arten von Herzversagen zu unterscheiden. Aktuell arbeitet er an einem Framework, mit dessen Hilfe Klinikerinnen und Kliniker bei der frühen Diagnose kardiovaskulärer Erkrankungen eine automatische Beurteilung der Herzmorphologie heranziehen können.
Herzmodelle für die klinische Praxis
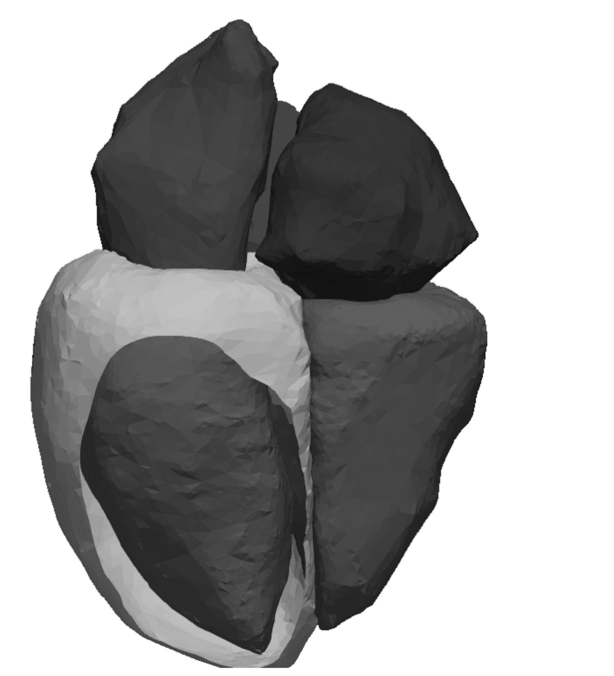
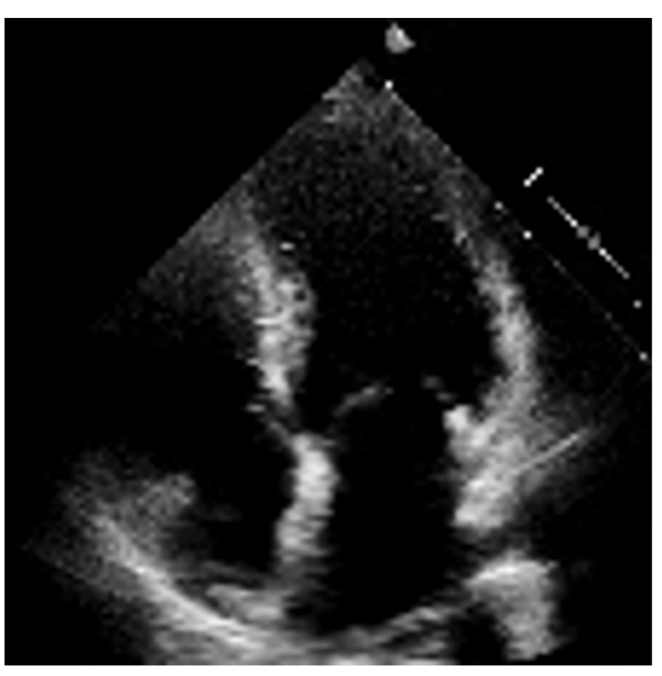
3D-Modelle des Herzens sind nicht neu, die existierenden Modelle basieren jedoch auf einer Magnetresonanztomografie (MRI) oder einer Computertomografie (CT). Dabei handelt es sich um sehr kostenintensive, bildgebende Verfahren. Diese sind für die Patient:innen bestenfalls unangenehm und werden nicht routinemässig durchgeführt. Im Gegensatz dazu sind Echokardiografien relativ kostengünstig, nicht invasiv und systematisch angewendet. Mittels 2D-Echokardiografie-Videos arbeitet Fabian an der Entwicklung eines neuen KI-Frameworks, welches ein vierdimensionales Modell der Herzform einer Patientin oder eines Patienten erstellt. Ausgehend davon kann man eine Vielzahl von Parametern ganz einfach mathematisch ableiten, unter anderem die linksventrikuläre Ejektionsfraktion. Diese Automatisierung kardialer Messungen erleichtert die Arbeit von Kliniker:innen bei der Erstellung von Untersuchungsberichten und wird hoffentlich dazu beitragen, die bestmögliche Behandlungsstrategie für jede Patientin und jeden Patienten festzulegen.
Das Modell verspricht darüber hinaus, neue Besonderheiten der Herzform und -bewegung erkennen zu lassen, durch die sich bestimmte Pathologien vorhersagen lassen könnten oder die mit bestimmten Resultaten in Verbindung stehen, wie beispielsweise der Wahrscheinlichkeit von Komplikationen oder dem Tod in den nächsten Monaten.
Herausforderungen bei der Nutzung von medizinischen Daten mittels maschinellem Lernen
Ein präzises, personalisiertes Modell der Herzform einer Patientin oder eines Patienten zu erstellen, ist jedoch komplizierter als es sich anhört. Obwohl in den vergangenen Jahren bedeutende Fortschritte beim maschinellen Lernen gemacht wurden, konnten sich im Gesundheitswesen nur sehr wenige Anwendungen erfolgreich durchsetzen. Dies ist nicht allein aufgrund der realen Herausforderungen beim Erfassen von Patientendaten und der ethischen Fragen im Zusammenhang mit der Technologienutzung in der Medizinbranche der Fall. Auch in Bezug auf medizinische Daten gibt es grosse technische Schwierigkeiten. Zum Beispiel bezüglich Heterogenität: Je nach Datenerheber:in, Patient:in und Bildgebungsgerät, mit dem die Daten erhoben wurden, variiert die Qualität der Daten erheblich. Bei einer Echokardiografie sind einige Videos kürzer, länger oder fokussieren auf unterschiedliche Teile des Herzens. Einige zoomen näher heran (oder heraus), beinhalten viele Geräusche oder wurden sogar in der Ambulanz auf dem Weg ins Spital gemacht.
Dies ist ein grosses Problem für Machine-Learning-Modelle, die in der Regel grosse Mengen an sauberen, homogenen Daten für ihr Training benötigen – insbesondere bei komplexen Datenformaten wie Videos. Grosse Mengen an qualitativ hochwertigen Echokardiografie-Videos sind jedoch nicht ohne Weiteres online verfügbar – im Gegensatz zu anderen Arten von Daten wie Tierbildern oder Text. Das Sammeln medizinischer Daten ist kompliziert, heikel und häufig durch Vorurteile belastet.

«Machine-Learning-Modelle lassen sich ausserhalb bestimmter Bereiche schlecht generalisieren. Eine Verschiebung des Bereichs kann zu ihrem kompletten Ausfall führen. Unser Modell sollte daher veränderten Rahmenbedingungen standhalten können: unterschiedlichen Kliniker:innen, Spitälern, Patientinnen und Patienten und so weiter…»Fabian Laumer, Doktorand in der Information and Science Engineering Group von Professor Buhmann
Die grösste technische Schwierigkeit für Fabian und seine Kolleginnen und Kollegen ergibt sich aus dem Fehlen einer sogenannten «Ground Truth». Dies ist das, was Informatikerinnen und Informatiker als die richtige oder "wahre" Antwort auf einen gegebenen Input bezeichnen. Es ist auch der Output, an dem das Machine-Learning-Modell lernen und validiert werden sollte. In diesem Fall ist dieser Output die endgültige 3D-Herzform.
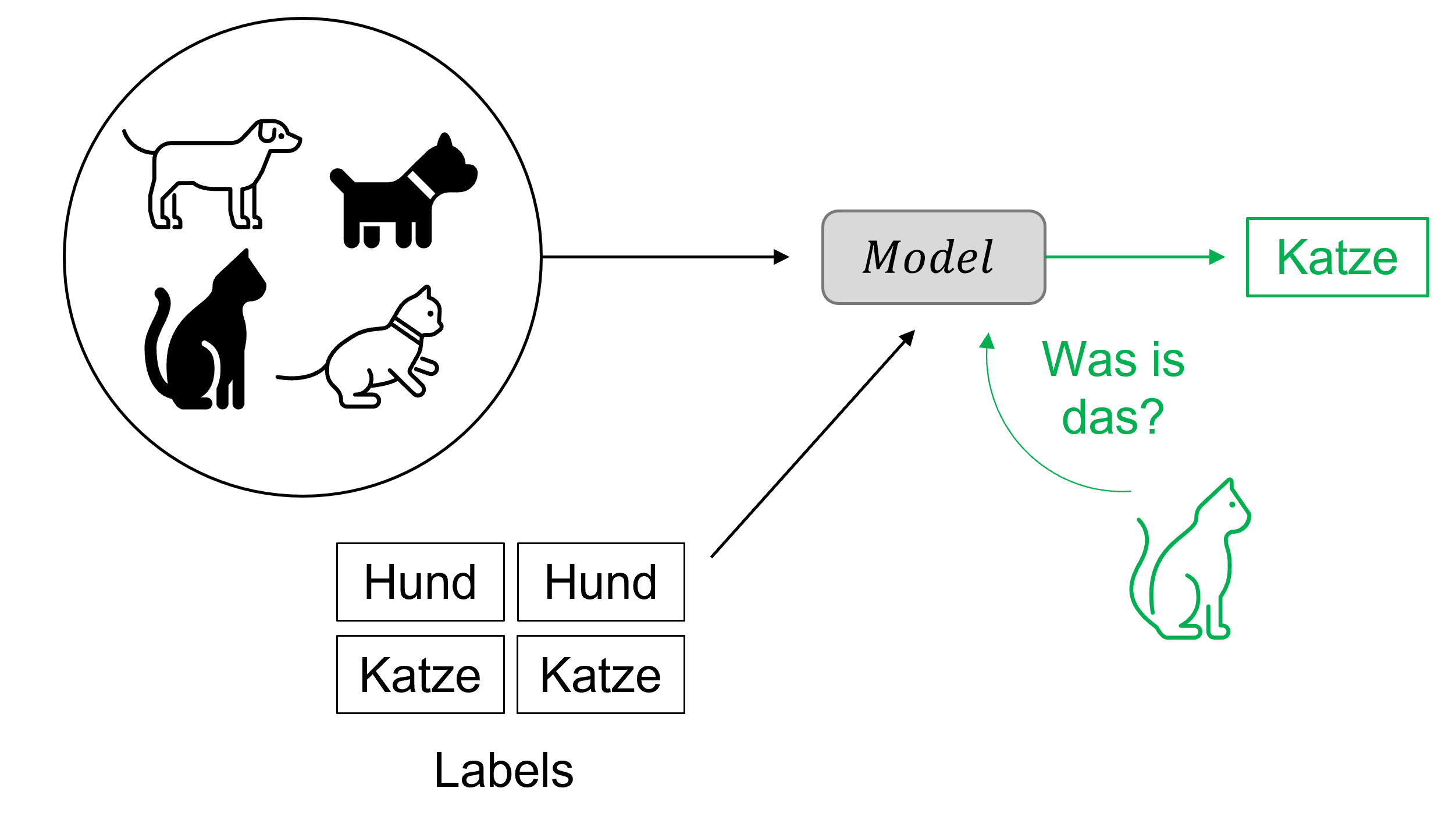
Klassische, überwachte Machine-Learning-Modelle nutzen gekennzeichnete Datensätze, um zu trainieren. Durch das Analysieren einer grossen Menge an mit «Katze» oder «Hund» gekennzeichneten Bildern lernt das Modell beispielsweise, eine Katze in einem neuen Datensatz mit nicht gekennzeichneten Bildern (von Katzen und Hunden) zu erkennen. Je mehr Bilder es im Laufe des Trainings zu sehen bekommt, desto besser wird es vorhersagen können, ob auf einem neuen Bild eine Katze zu sehen ist oder nicht. Fabian steht nun vor der Herausforderung, dass er keine gekennzeichneten Output-Daten hat. «Auch wenn wir den Input (das Echokardiografie-Video) haben», erklärt Fabian, «haben wir keine gekoppelten Input-Output-Daten, um das Modell zu trainieren.» Das entsprechende synthetische 3D-Herz (Output) für jede einzelne Patientin und jeden einzelnen Patienten existiert noch nicht.
Lernen ohne Ground Truth
Um das Fehlen von gepaarten Input-Output-Daten zu umgehen, entwickelte Fabian ein leicht überwachtes Machine-Learning-Modell, das einen physikalischen Prior verwendet. Zunächst generierte er einen Datensatz mit 10 000 synthetischen 3D-Herzformen mit Hilfe eines statistischen Formmodells, das aus einem Pool von CT-Bildern von etwa 20 gesunden Personen erstellt wurde.
Das Programm wird anschliessend trainiert, indem unterschiedliche Teile des Systems gegeneinander ins Rennen geschickt werden. Forschende, die sich mit maschinellem Lernen beschäftigen, nennen dies eine «cycleGAN-Architektur». Ein Teil (grau im Video) wird darauf trainiert, das eingegebene Echokardiografie-Video zu komprimieren, zu dekomprimieren und dann zu versuchen, das ursprüngliche Video zu rekonstruieren. Parallel hierzu führt ein zweiter Teil (hellblau im Video) die gleichen Abläufe mit den CT-basierten Videos vom synthetischen Herzen aus. Ein Diskriminator versucht kontinuierlich zwischen den Outputs, die vom Video einer tatsächlich Person stammen, und jenen, die auf ein synthetisches Herz zurückgehen, zu unterscheiden. Je besser der Diskriminator lernt, die Herkunft der Outputs zu unterscheiden, desto besser wird das Modell darin, diese zu generieren. Diese Konkurrenz führt zusammen mit der sogenannten «Zykluskonsistenz» (es sollte möglich sein, vom generierten personalisierten synthetischen Herzen aus zurück zum ursprünglichen Video zu gelangen) zur zunehmend besseren Qualität der erzeugten Ergebnisse.
Nach dem Training erzeugt das Programm ein synthetisches 3D-Herz, das personalisiert ist (da es vom Echokardiografie-Video einer Patientin oder eines Patienten stammt), jedoch auch der Verteilung der 3D-Herzformen, die aus CT-Bildern erstellt wurden, entspricht – d. h., es sieht aus, wie ein Herz in der Realität aussehen sollte. Sobald eine solche personalisierte, dynamische 3D-Form generiert worden ist, können die gewünschten klinischen Variablen, wie zum beispiel Ejektionsfraktion oder Ventrikelvolumen, ganz einfach extrahiert werden.

Eine weitere Herausforderung beim Fehlen gekennzeichneter Output-Daten ist die Beurteilung des Modells. Da es in der klinischen Praxis unterstützend genutzt wird, muss das Tool überaus verlässlich sein und die Ärzteschaft muss Fehler schnell erkennen können, statt blind darauf zu vertrauen. Es muss zudem robust sein und ungeachtet der unterschiedlichen Qualitäten der Input-Videos zuverlässig arbeiten. Ein Modell, das mit nur einigen sehr guten Videos perfekt arbeitet, wäre in der Praxis nicht sehr hilfreich. «Unser Modell muss immer gut genug arbeiten, statt nur manchmal perfekt zu funktionieren», fasst Fabian zusammen. Die Definition von «gut genug» in der klinischen Praxis ist und bleibt eine Herausforderung.
Bisher nutzten Fabian und seine Kolleginnen und Kollegen verschiedene Strategien, um die Leistung ihres Modells anzugleichen. Dies beinhaltete auch den Vergleich der computergenerierten, linksventrikulären Ejektionsfraktionen mit jenen, die durch die Ärzteschaft berechnet wurden – dem derzeitigen Goldstandard für diese Art von Messungen. Sie konnten ihr Modell auch anhand von zehn Patientinnen und Patienten testen, für die korrespondierende 3D-Formen auf Grundlage von MRI-Bildern verfügbar waren.
Ein Projekt in Zusammenarbeit mit Schweizer Spitälern
Um das Tool sowie verbesserte Versionen davon zu entwickeln, arbeiten Fabian und seine Kolleginnen und Kollegen mit vier Universitätsspitälern in Zürich, Bern, Genf und Basel sowie mit dem Swiss Heart Failure Network (SHFN) zusammen und haben bereits Daten von über 3000 Menschen gesammelt. Die Arbeit mit medizinischen Daten und Spitälern nimmt viel Zeit in Anspruch und ist manchmal herausfordernd. Die Chance, sich regelmässig mit Ärztinnen und Ärzten auszutauschen und die möglichen Auswirkungen ihrer Arbeit zu sehen, ist jedoch eine grosse Motivation für das Team. Sie treffen sich regelmässig mit Klinikerinnen und Klinikern, die äußerst interessiert und begeistert von der Technologie sind, die das Team entwickelt.
In zwei bis drei Jahren möchte Fabian das Tool in die medizinische Praxis einbringen. Die Umwandlung eines Forschungsprojekts in eine klinische Anwendung bedeutet viel Arbeit und beinhaltet ganz eigene Herausforderungen, kann jedoch seiner Ansicht nach in einer angemessenen Zeitspanne erfolgen. Die Software würde die Ärzteschaft in der Praxis zunächst beim Erstellen von Untersuchungsberichten und später auch unmittelbar bei diagnostischen Entscheidungen unterstützen. Für die Patientinnen und Patienten würden sich dadurch die Prognosen verbessern und es wären weniger invasive, klinische Untersuchungen und Behandlungen erforderlich.
Personen, die auch zum Projekt beigetragen haben:
-

Lena Rubi (Postdoktorandin im Labor von Prof. Joachim Buhmann) ist Teil der Zusammenarbeit mit dem SHFN und unterstützt das Projekt mit wertvollen Einblicken und Diskussionen. (Bild: D-INFK) -

Ami Beuret, ehemaliger Software-Ingenieur und Doktorand, hat bei der Implementierung der Software sehr geholfen, damit alles funktioniert. (Bild: D-INFK) -

Laura Manduchi (Doktorandin, Medical Data Science Group of Prof. Julia Vogt) ist Teil der Zusammenarbeit mit dem SHFN und unterstützt das Projekt mit wertvollen Einblicken und Diskussionen. (Bild: D-INFK) -

Alina Dubatovka (Doktorandin im Labor von Prof. Joachim Buhmann) ist Teil der Zusammenarbeit mit dem SHFN und unterstützt das Projekt mit wertvollen Einblicken und Diskussionen.
(Bild: D-INFK) -

Mounier Amrani hat das Projekt während seiner Masterarbeit unterstützt.
(Bild: D-INFK)
-

Dr. Med. Christian M. Matter (Kardiologe USZ) unterstützt das Team mit wertvollen Erkenntnissen aus medizinischer Sicht. -

Joachim M. Buhmann ist Professor und Leiter der Gruppe Information and Science Engineering (ISE) an der ETH Zürich.
(Bild: Giulia Marthaler)